Fabrication and application of double-layer nanopore
-
摘要: 由于具有孔径可调节、物理化学性质稳定、极端环境适应性强、集成度高等优点,固态纳米孔逐渐成为最具潜力的单分子测序工具。在固态纳米孔发展过程中,提高其单分子检测精度一直是研究人员关注的重点。近年来,双层纳米孔受到了广泛关注。与传统的单层纳米孔相比,双层纳米孔具有的孔-腔-孔结构提供了2个分子识别位点和纳米受限空间。双孔提供的2个分子识别位点可以在单次过孔事件中获得2次目标信号,所获得的双重检测信号不仅丰富了检测信息,也为信号分析提供了最为直接的对比信息源。此外,双层孔中的空腔还可作为单分子化学反应器。因此,双层纳米孔的出现拓宽了纳米孔传感器的应用范围,在单分子检测方面具有广阔的应用前景。本文概述了纳米孔的发展历程,并重点介绍了双层纳米孔的制造方法及其在单分子检测领域的应用。Abstract: Solid-state nanopores have become the most promising single-molecule sequencing tools due to their adjustable pore size, stable physical and chemical properties, strong adaptability to extreme environments, and high integration. In the area of solid-state nanopores research, improving the detection accuracy of single molecules has always been the core concern of researchers. In recent years, the double-layer nanopores has attracted wide attention. Compared with traditional single-layer nanopore, the "pore-cavity-pore" structure of double-layer nanopores provides two molecular recognition sites and nanoconfined space. The two molecular recognition sites provided by the two nanopores can obtain two target signals in a single translocation event, which not only enrich the detection information but also provide the most direct comparison information source for signal analysis. In addition, the "cavity" in the double-layer nanopores can be used as a single-molecule chemical reactor. Therefore, the appearance of double-layer nanopores broadens the application range of nanopore sensors and has an important application prospect in single molecule detection. In this paper, the development history of nanopores is summarized, and the fabrication methods of double-layer nanopores and their applications in the field of single-molecule detection are emphatically introduced.
-
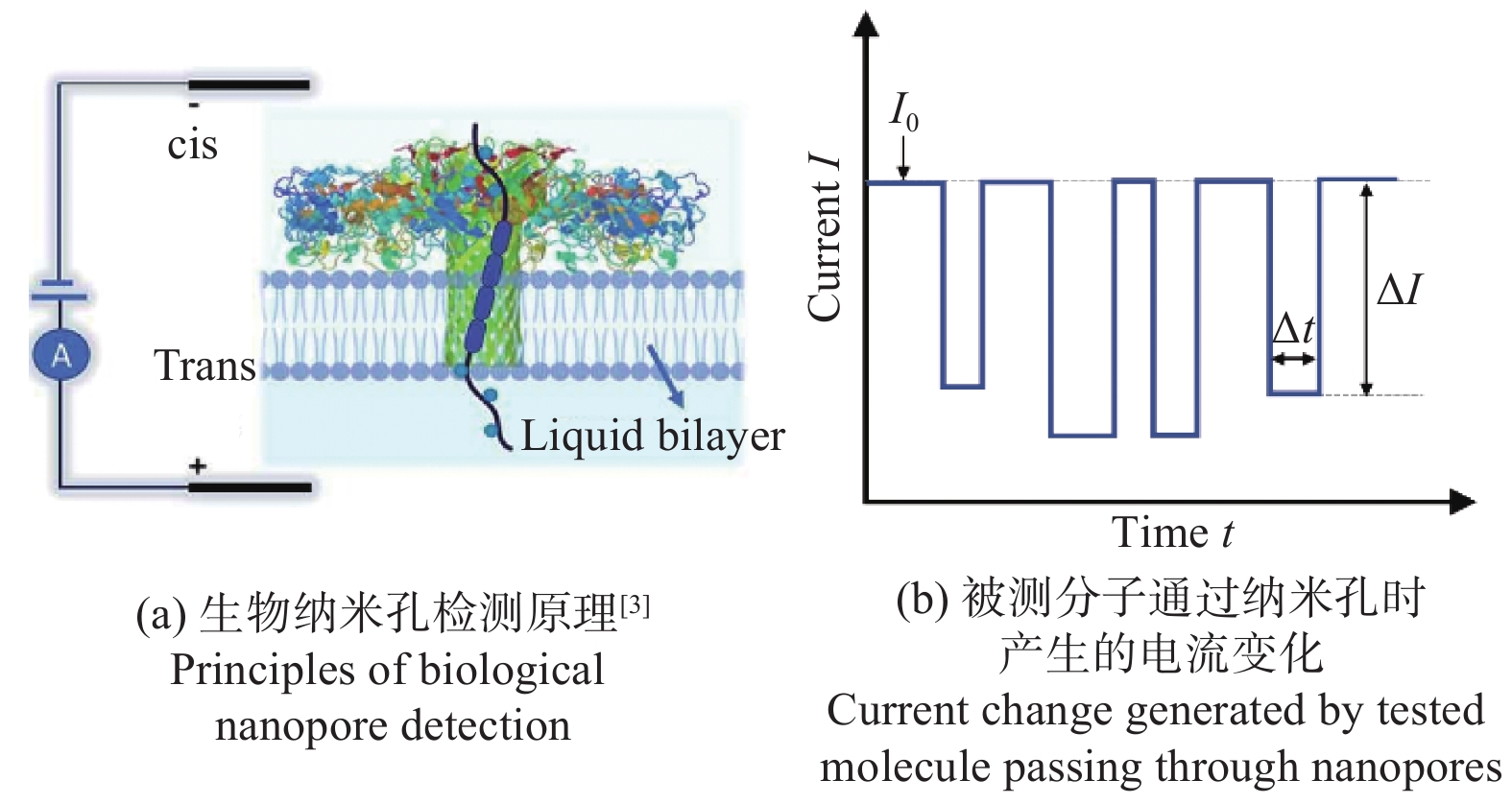
纳米孔检测的应用最早可以追溯到19世纪50年代库尔特计数器的发明[1]。如图1所示,充满电解质溶液的液池被纳米孔薄膜分隔成2个独立的腔室,电压施加在两端腔室上,此时溶液中离子运动形成稳定电流I0。向溶液一侧加入被测分子后,被测分子将在电场力的作用下从一端腔室经过纳米孔运动到另一端腔室。被测分子在孔内的物理占位使离子发生扰动,从而产生电流变化∆I。通过分析电流变化的幅值、持续时间、频率等,可以获取被测分子的大小、浓度、电荷和构象等信息。
KASIANOWICZ等[2]首次使用α-溶血素生物纳米孔成功检测到单链脱氧核糖核酸(ssDNA)易位产生的离子电流信号,由此拉开了纳米孔单分子检测的序幕。自此,纳米孔凭借其免标记、长读长、实时检测等优点,在核酸和蛋白质测序、生物分子检测和纳米颗粒表征等方面得到了广泛应用,并成为最具有发展前景的单分子传感器之一。
根据材料的不同,纳米孔主要分为生物纳米孔和固态纳米孔[3]。生物纳米孔是由特定的氨基酸序列自组装形成的天然通道蛋白,通常需要将其嵌入脂质双分子层或合成膜中构成生物纳米孔传感器。最具代表性的生物纳米孔有α-溶血素[4]、MspA[5]、phi29[6]等,这些纳米孔已广泛用于单分子检测。由于具有孔径定义明确、信号再现性强、噪声水平低等优点,生物纳米孔目前已在核酸测序方面得到了成功应用[7-8],且随着纳米孔化学、碱基调用算法和数据校正算法的更新,其读数准确性已经从低于60%提高到99%以上[9],具有临床应用前景,如监测传染病疫情、表征癌症的结构变体等[10-12]。生物纳米孔检测为防范病毒感染,保障人类生命健康提供了可靠的检测手段。然而,使用寿命短、极端环境耐受性差等缺点限制了生物纳米孔的进一步应用。
与生物纳米孔相比,固态纳米孔具有孔径可调节、物理化学性质稳定、极端环境适应性强、集成度高等优点,因而备受关注。LI等[13]首次使用Ar + 离子束在单层悬浮氮化硅薄膜上制备了单个纳米孔,并将其成功用于DNA检测。随后,基于透射电子显微镜的高能电子束固态纳米孔原位制造与表征技术出现[14],固态纳米孔开始得到广泛研究。然而,较低的单分子检测精度制约了固态纳米孔在单分子检测方面的应用。得益于微纳加工技术的发展,镓/氦离子聚焦离子束[15-16]、电击穿[17]等纳米孔制造技术逐步发展,从而将纳米孔的孔径控制在与单分子尺寸相当的范围内。材料科学的发展也促使制造固态纳米孔的材料从氮化硅[18-20]、二氧化硅[14]、二氧化铪[21]等半导体材料拓展到石墨烯、二硫化钼、二硫化钨等二维材料。这些二维材料的厚度与DNA相邻碱基的间距相当,大大提高了纳米孔对单碱基的空间分辨率[22-23]。
除了从纳米孔的孔径和厚度等方面提高检测精度,光镊、磁镊、音叉和AFM探针等工具也被用于控制单分子经过纳米孔的易位速度,并通过操纵单分子在纳米孔内往复运动来实现电流信号的多次读取,从而提高检测精度[24]。得益于固态纳米孔与半导体工艺的高度兼容性,人们开发出纳米孔-隧穿结[25-26]、纳米孔-场效应管[27-28]、等离子体纳米孔[29]等集成结构,这些结构在传统离子电流检测模式的基础上增加了隧穿电流、横向电流、光谱等检测模式,因此可以通过组合多种模式来提高纳米孔的检测精度。
提高检测精度的另一种方法是制造具有多个串联纳米孔的纳米流体结构。近年来,双层纳米孔受到了研究人员的广泛关注。与传统的单层纳米孔相比,双层纳米孔具有的孔-腔-孔结构提供了2个分子识别位点和纳米受限空间。双孔提供的2个分子识别位点可以在单次过孔事件中获得2次目标信号。通过分析2次目标信号的时间间隔或形状差异,可以推算分子的电泳飞行时间和Zeta电位[30],或用于DNA动力学校对实现DNA测序[31]。此外,双层孔中的空腔还可作为单分子化学反应器[32]。因此,双层纳米孔的出现拓宽了纳米孔传感器的应用范围,在单分子检测方面具有广阔的应用前景。本文概述了纳米孔的发展历程,并从其在单分子检测方面的应用出发,重点介绍了双层纳米孔的制造方法。
1. 双层纳米孔的制造方法
双层纳米孔的制造是基于PEDONE等[33]提出的堆叠孔-腔-孔结构(图2)。该结构是由氮化硅纳米孔、硅空腔、硅纳米孔依次在垂直方向上堆叠形成的。整个结构的制造主要分为2个步骤:首先,通过光学光刻、电子束光刻与反应离子刻蚀等步骤在氮化硅层中加工第1个纳米孔,即氮化硅纳米孔,如图2a所示;然后,通过反馈控制湿法化学刻蚀在硅层中加工第2个纳米孔,即硅纳米孔,如图2b所示。由于硅在碱性溶液中的各向异性湿法刻蚀作用,这种方法加工出来的双层孔的空腔形状几乎为锥形。空腔的大小通过控制刻蚀溶液的刻蚀时间来调节,而氮化硅纳米孔和硅纳米孔的大小则分别通过调整电子束曝光的剂量以及反馈湿法化学刻蚀中的电流阈值来控制。通过该方法获得的孔-腔-孔结构中,氮化硅孔直径为28 nm,硅孔边长为23 nm × 23 nm,空腔高度为1.5 μm,如图2c所示。该方法所制造的硅纳米孔大多呈矩形。
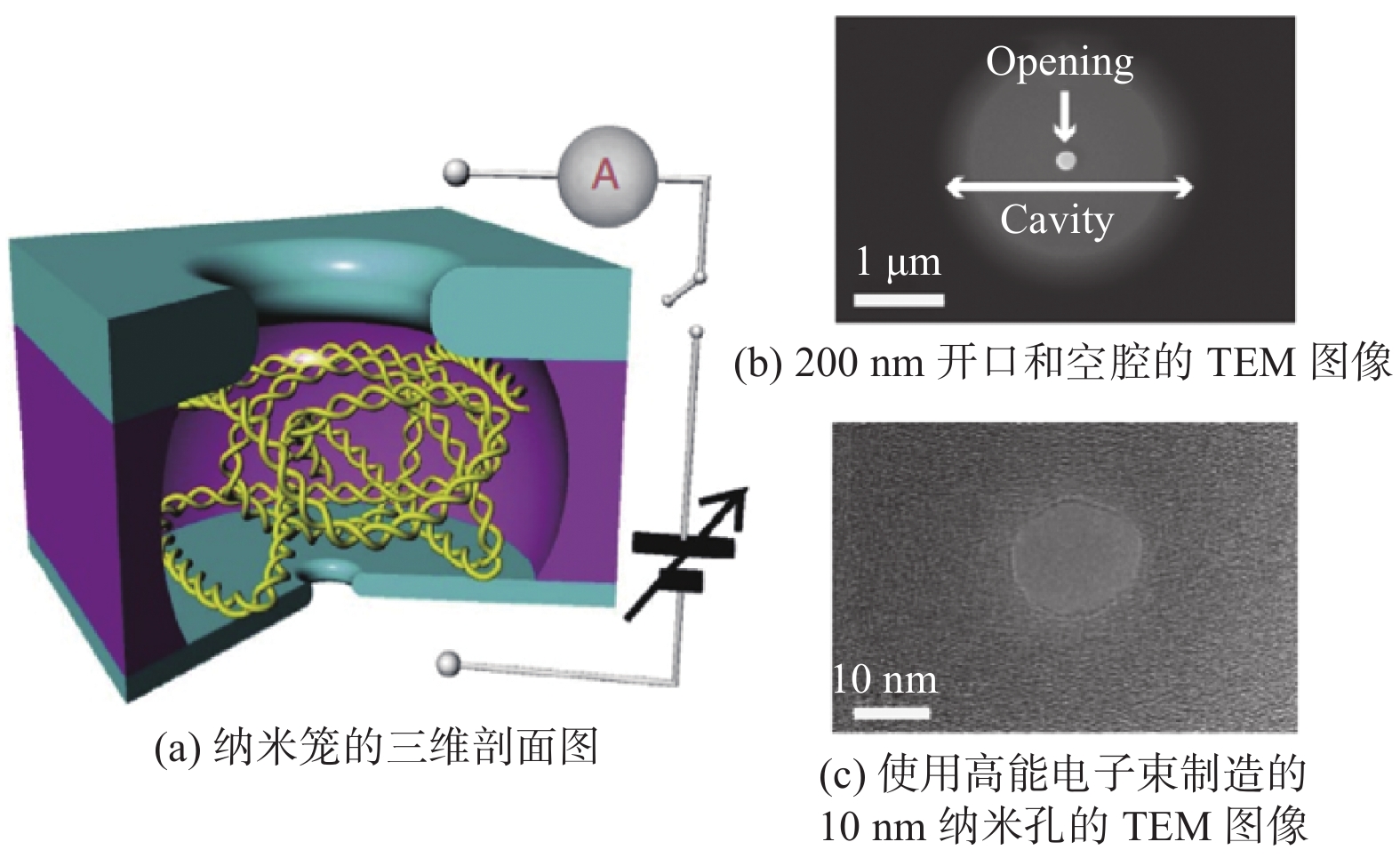
LIU等[32]制造了一种纳米孔熵笼的双层孔结构,如图3所示。首先,选取硅基底上的自支撑悬浮薄膜(自上而下依次为20 nm厚的氮化硅层、1.5 mm厚的二氧化硅层以及400 nm厚的氮化硅层)。然后,使用聚集离子束在400 nm厚的氮化硅层上加工出直径为200 nm的开口。接着,利用氢氟酸溶液腐蚀中间的二氧化硅,得到1.5 mm高的圆柱形空腔。最后,利用透射电子显微镜的电子束在20 nm厚的氮化硅薄膜上加工10 nm的纳米孔。其中,空腔的大小通过控制氢氟酸的刻蚀时间来调节,纳米孔的大小则通过调整聚焦离子束和高能电子束的加工参数来控制。此方法极大地简化了制造流程,且可控性更强。由于该方法利用了TEM原位制造技术,在纳米孔制造完成后还可原位获得高分辨TEM图像。叶佳佳[34]讨论了基于镓离子束和氦离子束的双层孔制造流程及其TEM无损表征方法。在双层孔制造完成后,利用TEM的高分辨成像和能谱分析功能完整表征了双层孔的三维结构,验证双层孔成功制造的同时保证了该结构的完整性。
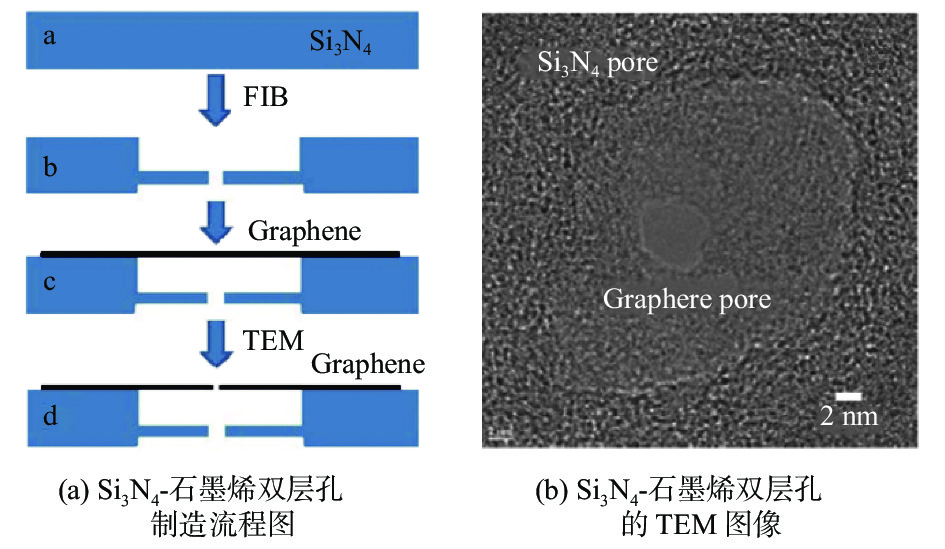
SiO2和Si3N4薄膜在固态纳米孔中的广泛应用得益于其较高的化学稳定性和较低的机械应力。随着材料科学的发展,石墨烯、二硫化钼、二硫化钨等二维材料由于具有原子级厚度和良好的机械稳定性也被广泛用于固态纳米孔的制造中。石墨烯纳米孔与Si3N4纳米孔亦可组合成双层孔(图4)[35]。首先,使用聚焦离子束将低应力Si3N4薄膜(初始厚度为100 nm)减薄至15 nm;然后,利用FIB在15 nm的Si3N4薄膜上加工,得到直径为28 nm的Si3N4纳米孔;接着,通过湿法转移技术,将石墨烯薄膜转移到上述Si3N4薄膜的顶部,将Si3N4纳米孔覆盖;最后,利用TEM在石墨烯薄膜上加工,得到4 nm的石墨烯纳米孔。这种方法制造的双层孔结构的空腔和纳米孔大小均由聚焦离子束和高能电子束的加工参数来调节,具有较高的可控性。
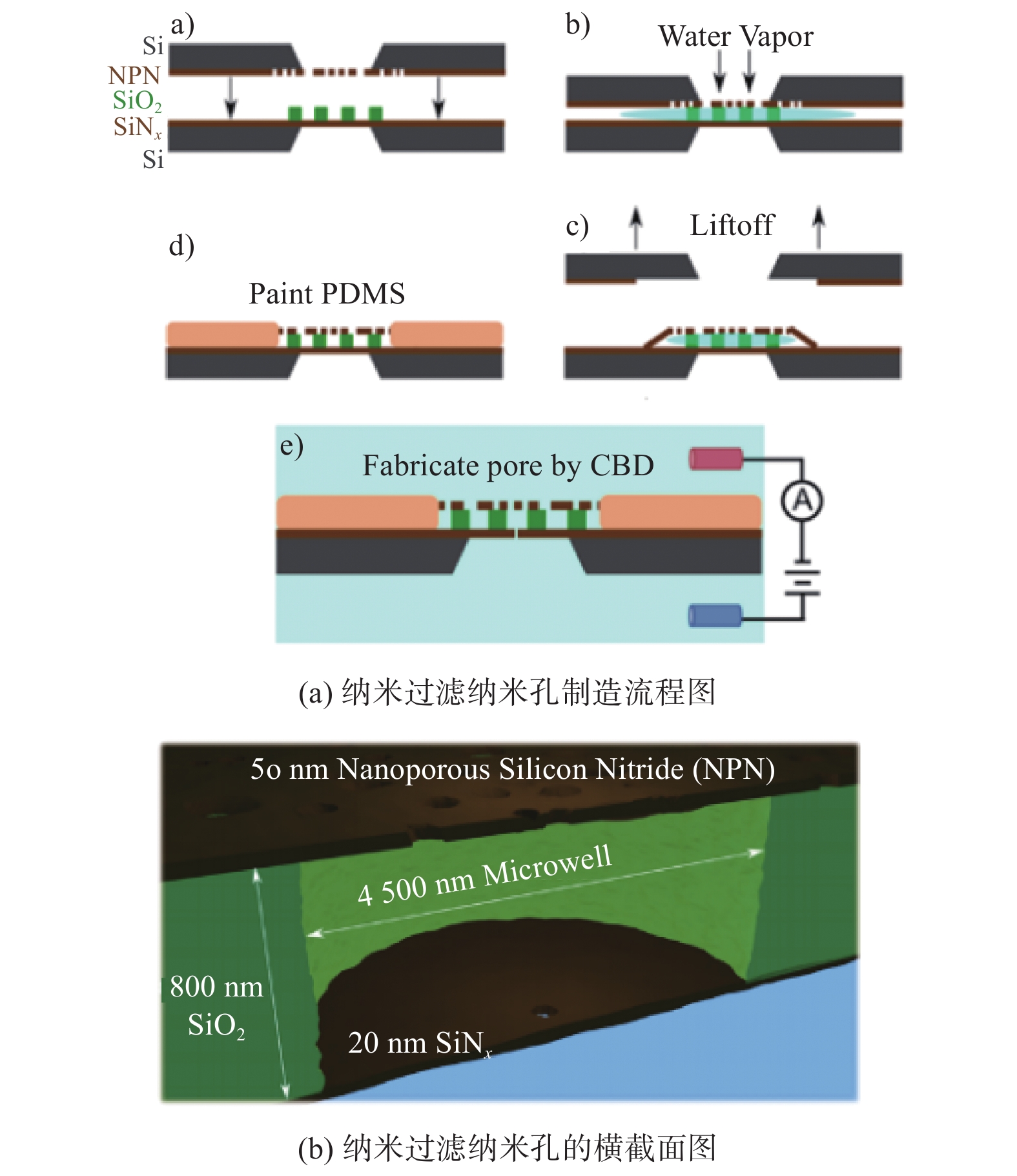
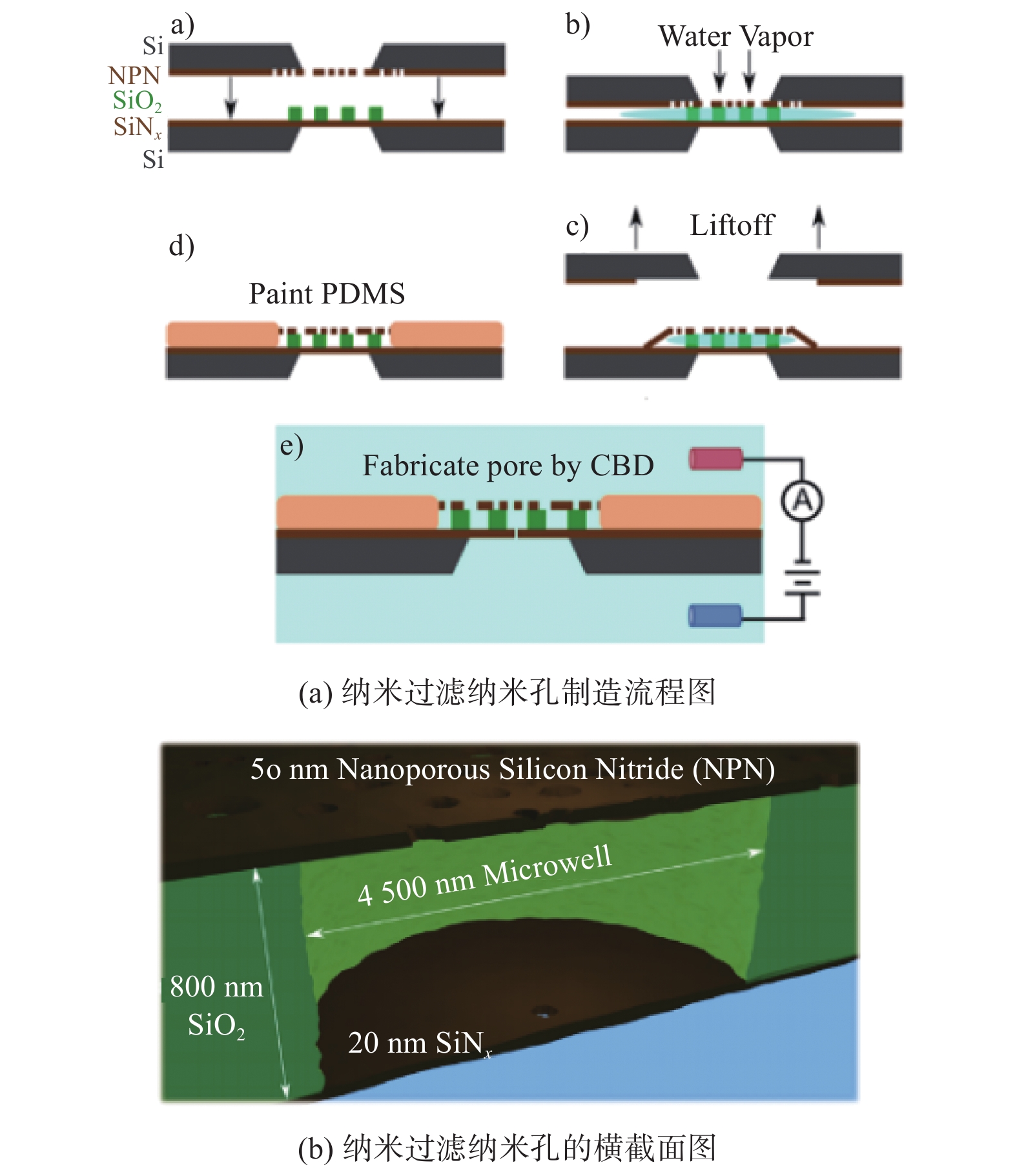
LAM等[36]制造了纳米孔阵列膜-纳米孔结构,如图5所示。这种双层孔结构的上层为纳米孔阵列膜,下层为单个纳米孔。其制备过程包括3个环节:首先使用水蒸气分层法和lift off技术,将50 nm厚的纳米孔阵列膜附着在具有二氧化硅间隔层的20 nm厚的氮化硅薄膜上,其中二氧化硅间隔层内含有直径为4.5 μm的微孔六边形网格;接着,将PDMS涂覆在氮化硅薄膜和纳米过滤膜的边界上,使2层薄膜牢固结合;最后,使用电击穿方法,在20 nm厚的氮化硅薄膜上加工直径为6~15 nm的纳米孔。最终得到的纳米孔阵列膜-纳米孔结构如图5b所示。
2. 双层纳米孔的应用
与传统的单层纳米孔相比,双层纳米孔的孔-腔-孔结构提供了2个独立的分子识别位点,因此可以对同一被测分子进行2次连续的电流信号检测。通过分析2次电流信号的时间间隔和形状差异,可以推算被测分子的电泳飞行时间[30],或用于DNA动力学校对[31]。目前,双层纳米孔已在核酸分子、纳米颗粒的研究中得到了成功应用。孔-腔-孔结构按照排列方式及检测原理的不同,主要可以分为堆叠孔-腔-孔和平面孔-腔-孔。
2.1 堆叠孔-腔-孔
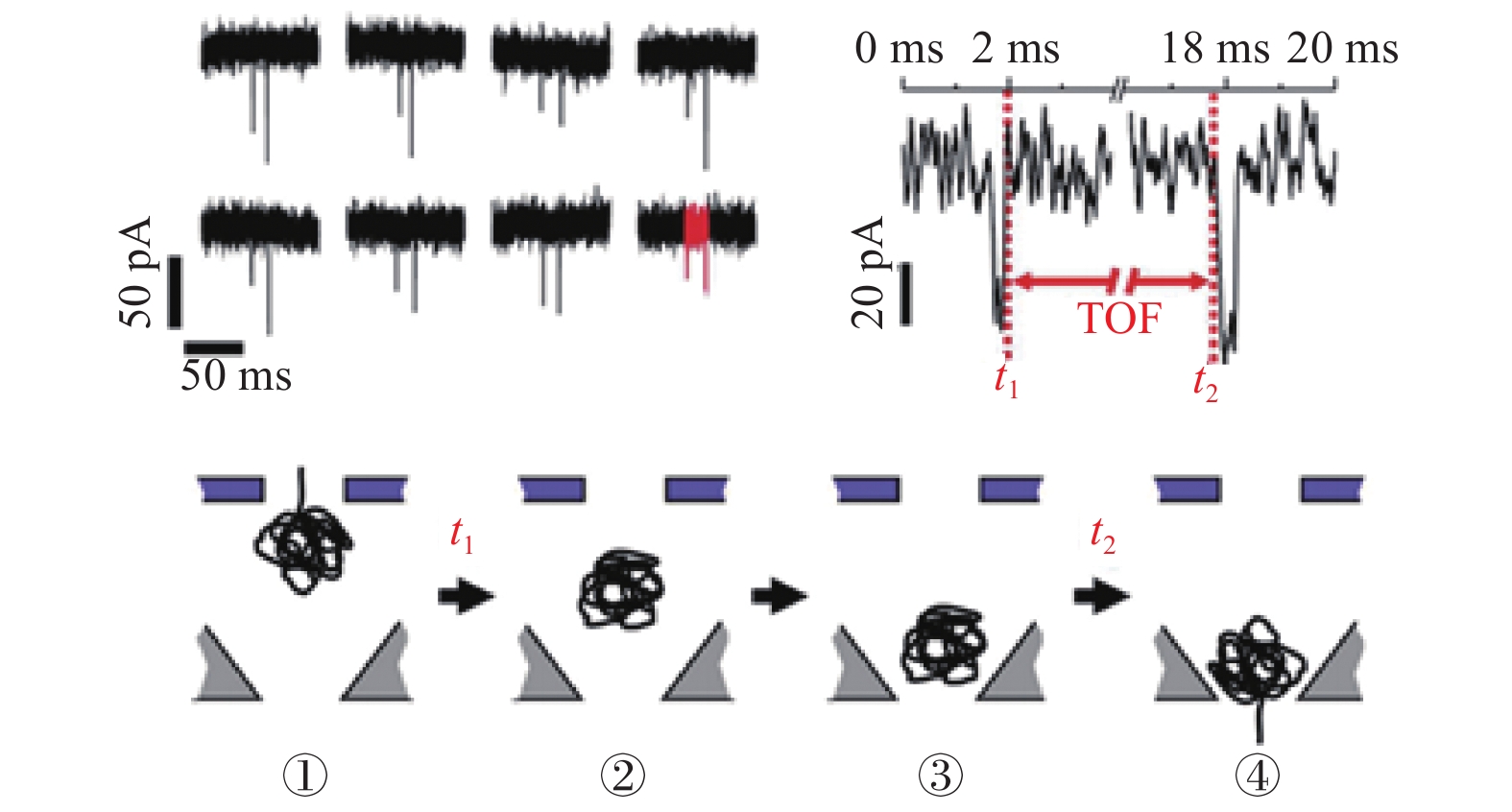
图6所示为LANGECKER等[30]使用堆叠孔-腔-孔结构研究λDNA分子的电泳飞行时间的示意图。利用空腔距离为1.5 μm的堆叠孔-腔-孔装置来测量λDNA分子在自由溶液中的电泳迁移率。在通过实验得到的电流轨迹线中,观察到一系列的电流峰,这些电流峰归因于单个分子通过孔-腔-孔结构中的1个纳米孔易位。因此,每个纳米孔可以独立作为传统的电阻脉冲传感器工作。DNA分子在电场驱动下通过会依次经过2个纳米孔,因此电流峰是成对的。测量并统计每个电流峰对中2个电流峰的距离,可以获得单个DNA分子的飞行时间(time-of-flight,TOF),从而可以在极低的浓度下确定DNA分子的电泳迁移率、Zeta电位,以及漂移、扩散和势垒主导的逃逸对DNA通过PCP装置易位的影响。随后,孔-腔-孔结构被用于研究单个分子在受限空间下的捕获、逃逸和扩散行为。通过调整电位极性,最终实现10 fL体积的空腔内带负电颗粒的捕获、储存和驱逐[37]。
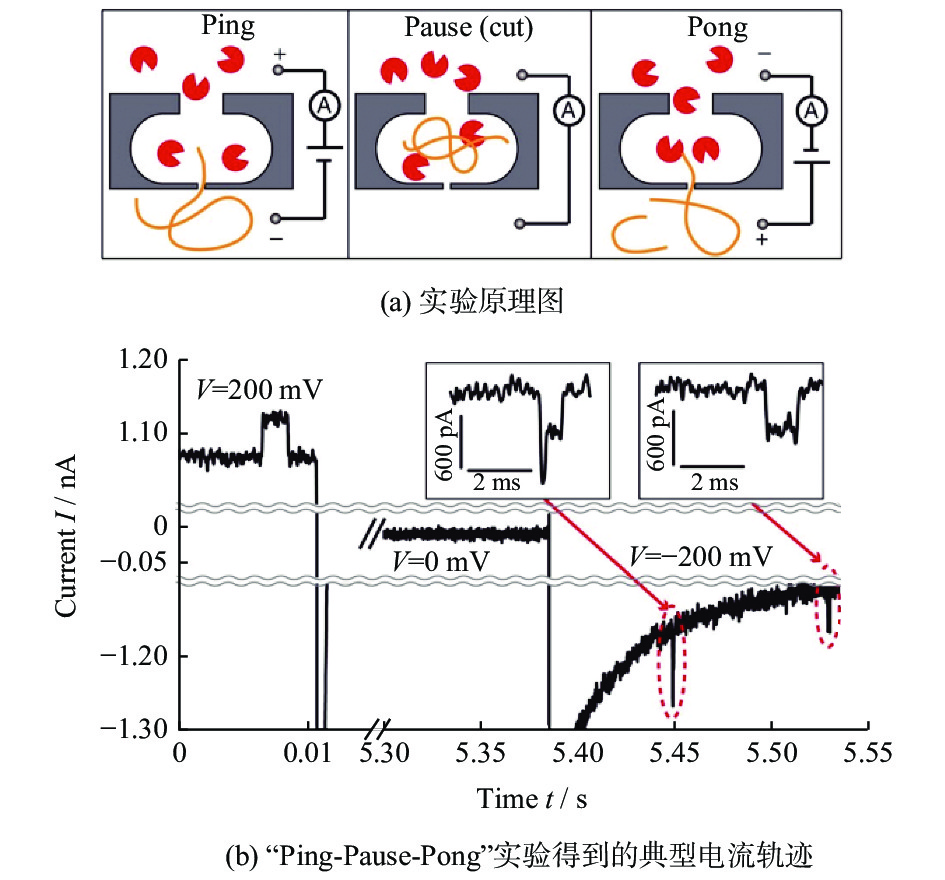
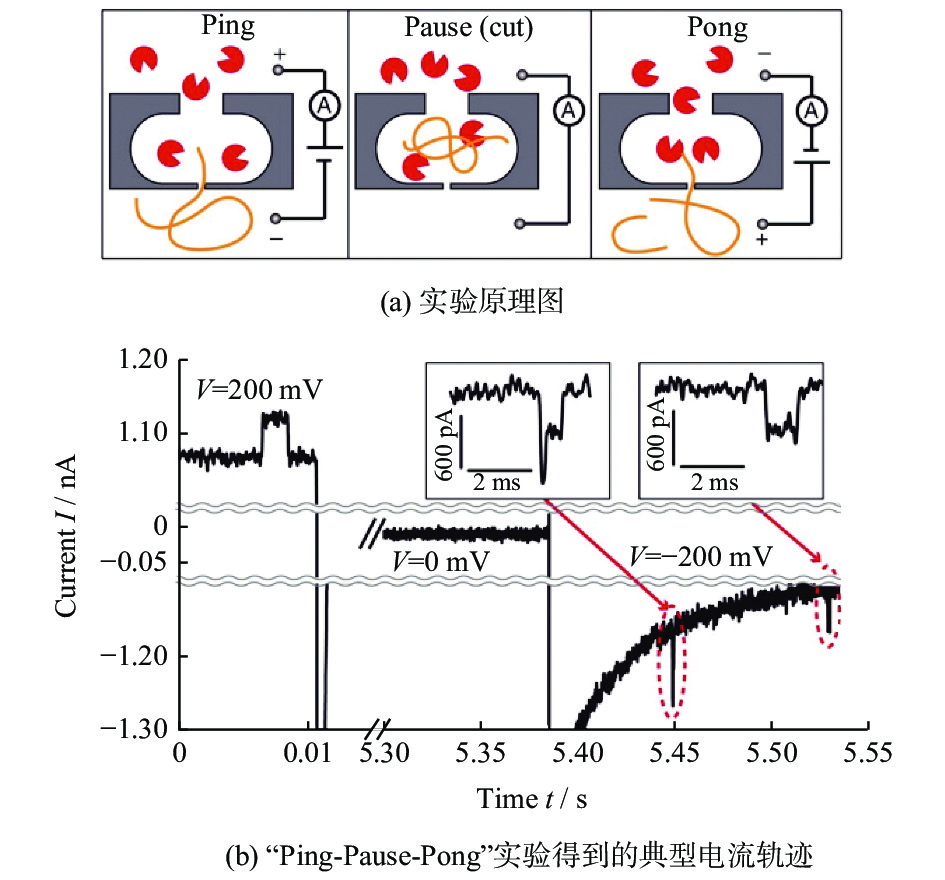
堆叠孔-腔-孔结构还可以作为单分子化学反应器(纳米熵笼),研究其在化学反应前后的变化[32]。纳米熵笼单分子化学反应器如图7所示。正极腔室中为含有限制性内切酶的缓冲液,负极腔室中为含有λDNA分子但不含酶的相同缓冲液。首先施加正电压将λDNA分子通过纳米孔拉入熵笼中,此过程产生的电流变化称为Ping。电流信号向上是由于DNA携带了大量的反离子进入纳米孔,使溶液的电导率增加。随后,将电压调零,此时λDNA失去外力作用而停留在熵笼中(这一过程称为Pause),并在熵笼中与限制性内切酶反应。最后反转电压极性为负电压,将DNA逐出纳米孔,此时产生的电流变化称为Pong。从图7b可知,1个Ping产生了2个Pong,说明原本完整的λDNA分子被切割形成了2个较短的DNA片段。纳米熵笼结构为研究体外模拟噬菌体耐药机制的限制性修饰系统、限制性片段长度多态性的遗传变异、实时研究DNA酶切提供了快速、免标记的方法。
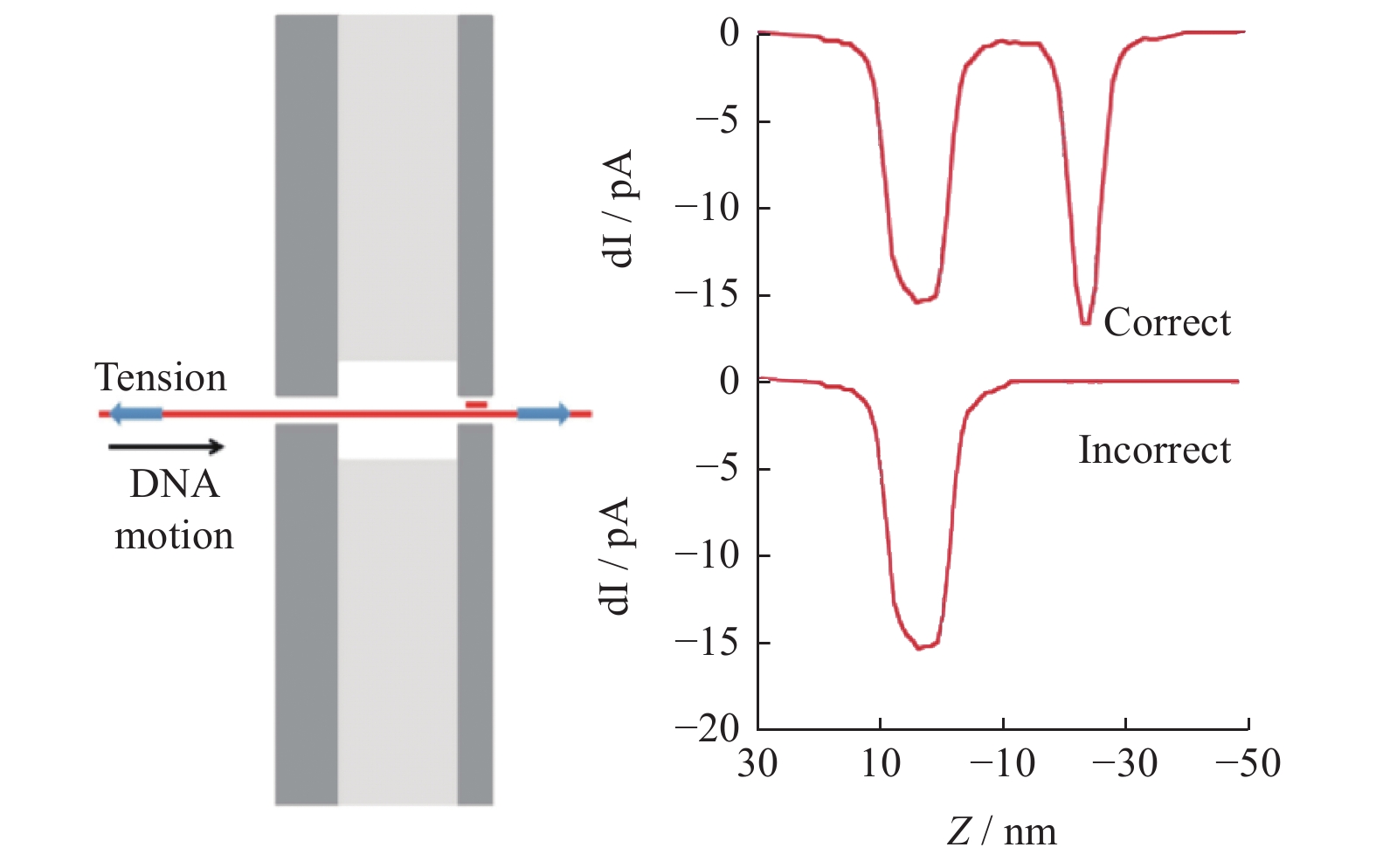
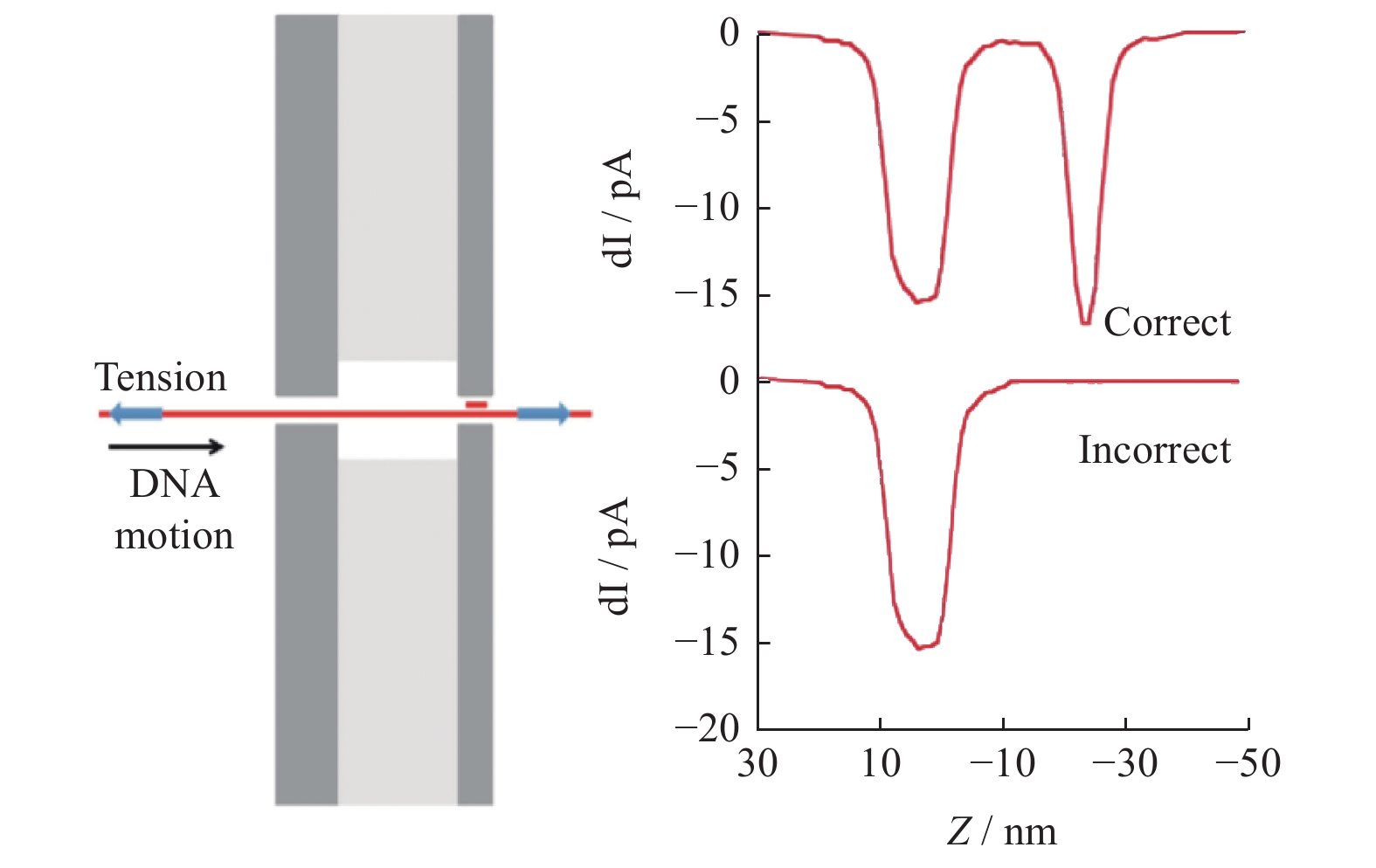
双层孔提供的校对机制有助于提高基因测序的精度。LING等[31]提出使用双层孔结合寡核苷酸动力学原理进行DNA测序。如图8所示,ssDNA被电压驱动进入双层孔结构,随后向双层孔的其中一端加入寡核苷酸探针,探针通过碱基配对原则与ssDNA结合。若该探针在ssDNA上是正确配对的,则DNA连续通过2个纳米孔时,会由于探针的出现而产生2次电流下降。若该探针不是正确配对的,则探针会在到达第2个孔之前从ssDNA上分离。这是因为仅有几个碱基的寡核苷酸探针具有较大的扩散常数,会很快被电压驱逐,降低重新杂交的可能性,所以在过孔电流中只能观察到第1次电流下降。这种方法避免了传统测序方法中DNA聚合酶的使用,使测序过程简化,同时在一定程度上缩短了测序时间,提高了检测精度。
2.2 平面孔-腔-孔
平面孔-腔-孔的结构类似于堆叠孔-腔-孔结构,2个独立的纳米通道提供了2个分子识别位点,因此可以获得2次电流信号变化,从而推算被测分子的电泳迁移率或提高检测精度,其应用如图9所示。
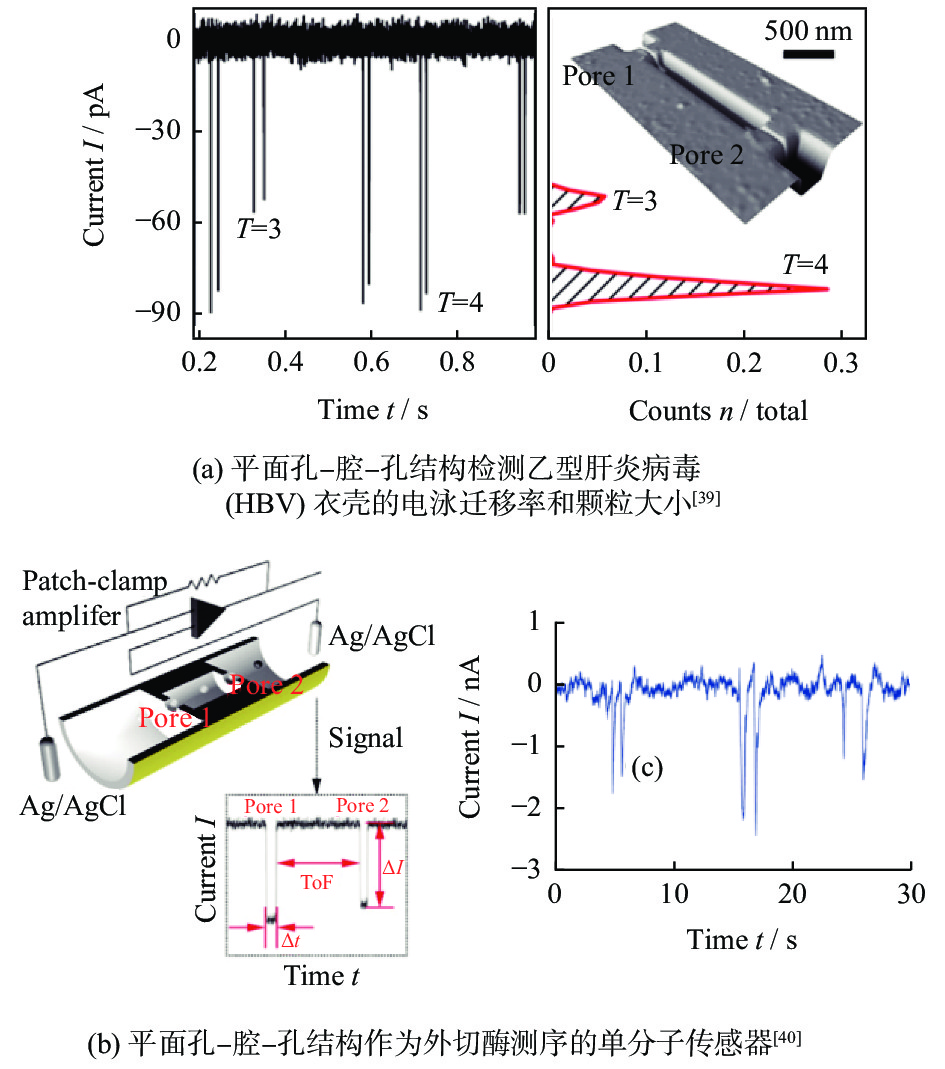
HARMS等[38]利用孔-孔间距为2.5 μm的平面孔-腔-孔装置检测乙型肝炎病毒(HBV),获得了一系列双电阻脉冲事件(图9a),通过分析双电阻脉冲之间的距离以及单个脉冲的幅值和宽度,计算出HBV的电泳迁移率,并实现了T=3(32 nm)和T=4(35 nm)时2种HBV病毒的识别。孔-腔-孔纳米传感器可以与DNA/RNA外切酶相结合,实现高读取精度的外切酶测序。CHOI等[39-40]利用腔体长度为5 μm的平面孔-腔-孔传感器鉴定了4种2-脱氧核糖核苷-5'-单磷酸(dNMPs分子)(图9b)。识别精度与通道的长度有关,最高可达94%。实验发现,不同dNMPs分子过孔形成的峰值对间距(TOF)不同,因此可以判断出dNMPs分子的种类。为了确定与相邻峰对完全独立的峰对,制定了峰对识别的标准:(1)2个电流峰的幅值应至少为通孔电流噪声均方根值(I-RMS)的3倍;(2)TOF的最小值应大于单个电流峰的持续时间;(3)TOF的最大值应为dNMPs分子在纳米电泳中的最小迁移率TOF计算值的1.5倍。部分未成对的峰归因于以下2点:(1)峰缺失——同一dNMPs分子在纳米孔不同位置易位导致电流峰发生改变,或是受限于电流放大器的带宽导致无法记录个别dNMPs分子的快速易位;(2)峰重叠——dNMPs分子的浓度过高,导致多个分子同时过孔。
3. 结论
目前,双层孔主要由电子束光刻、湿法刻蚀、聚焦离子束、透射电子显微镜和电击穿等中的2种或3种方法组合制造而成。不同的制造方法在加工效率、加工精度、加工成本和可控性等方面各有优缺点。例如,电击穿法加工精度高且成本低,但孔的数量和位置较难控制;电子束光刻法可控性较高,但其工艺成本昂贵;反馈控制湿法化学刻蚀成本较低但可控性较低;聚焦离子束和透射电子显微镜的电子束具有加工精度高、可控性高的优点,但成本较高。其中,聚焦离子束相对透射电子显微镜的加工效率更高。在实际加工过程中,应根据实际合理选择制造方法,提高双层孔尺寸和几何形状的可控性,使其与分析物的大小相匹配,获得良好的信号再现性和最佳的检测信噪比。在检测方面,双层孔目前已用于研究分子的电泳飞行时间、分子在受限空间中的行为、单分子化学反应以及提高纳米孔的检测精度。
随着微纳加工技术、材料科学和算法工具的快速发展,未来将有望开发更多识别位点的纳米串联多孔结构,从而在原始数据获取和后期数据分析过程中提高纳米孔的读数准确性,获得高质量的检测信号,实现真正简单、快速、便携、可靠、精准的纳米孔检测方案,加快纳米孔传感器在临床应用中的转化,推动精准医疗的发展。
-
-
[1] COULTER W H. Means for counting particles suspended in a fluid: US11281949A [P]. 1953-10-20. [2] KASIANOWICZ J J, BRANDIN E, BRANTON D, et al. Characterization of individual polynucleotide molecules using a membrane channel [J]. Proceedings of the National Academy of Sciences of the United States of America,1996,93(24):13770-13773. doi: 10.1073/pnas.93.24.13770 [3] AKHTARIAN S, MIRI S, DOOSTMOHAMMADI A, et al. Nanopore sensors for viral particle quantification: current progress and future prospects [J]. Bioengineered,2021,12(2):9189-9215. doi: 10.1080/21655979.2021.1995991 [4] CLARKE J, WU H C, JAYASINGHE L, et al. Continuous base identification for single-molecule nanopore DNA sequencing [J]. Nature Nanotechnology,2009,4(4):265-270. doi: 10.1038/nnano.2009.12 [5] MANRAO E A, DERRINGTON I M, LASZLO A H, et al. Reading DNA at single-nucleotide resolution with a mutant MspA nanopore and phi29 DNA polymerase [J]. Nature Biotechnology,2012,30(4):349-353. doi: 10.1038/nbt.2171 [6] WENDELL D, JING P, GENG J, et al. Translocation of double-stranded DNA through membrane-adapted phi29 motor protein nanopores [J]. Nature Nanotechnology,2009,4(11):765-772. doi: 10.1038/nnano.2009.259 [7] MULLEY J F, HARGREAVES A D. Assessing the utility of the Oxford Nanopore MinION for snake venom gland cDNA sequencing [J]. Peerj,2015,3(3):e1441. doi: 10.7717/peerj.1441 [8] JAIN M, OLSEN H E, PATEN B, et al. The Oxford nanopore MinION: delivery of nanopore sequencing to the genomics community [J]. Genome Biology,2016,17(1):239. doi: 10.1186/s13059-016-1103-0 [9] RANG F J, KLOOSTERMAN W P, JEROEN D R. From squiggle to basepair: computational approaches for improving nanopore sequencing read accuracy [J]. Genome Biology,2018,19(1):90. doi: 10.1186/s13059-018-1462-9 [10] BOZA V, BREJOVA B, VINAR T. DeepNano: Deep recurrent neural networks for base calling in MinION Nanopore reads [J]. PLoS ONE,2017,12(6):e0178751. doi: 10.1371/journal.pone.0178751 [11] LIN B, HUI J, MAO H. Nanopore technology and its applications in gene sequencing [J]. Biosensors,2021,11(7):214. doi: 10.3390/bios11070214 [12] BULL R A, ADIKARI T N, FERGUSON J M, et al. Analytical validity of nanopore sequencing for rapid SARS-CoV-2 genome analysis [J]. Nature Communications,2020,11(1):6272. doi: 10.1038/s41467-019-13858-z [13] LI J, STEIN D, MCMULLAN C, et al. Ion-beam sculpting at nanometre length scales [J]. Nature,2001,412(6843):166-169. doi: 10.1038/35084037 [14] STORM A J, CHEN J H, LING X S, et al. Fabrication of solid-state nanopores with single-nanometre precision [J]. Nature Materials,2003,2(8):537-540. doi: 10.1038/nmat941 [15] GIERAK J, MADOURI A, BIANCE A L, et al. Sub-5 nm FIB direct patterning of nanodevices [J]. Microelectronic Engineering,2007,84(5/6/7/8):779-783. doi: 10.1016/j.mee.2007.01.059 [16] YANG J, FERRANTI D C, STERN L A, et al. Rapid and precise scanning helium ion microscope milling of solid-state nanopores for biomolecule detection [J]. Nanotechnology,2011,22(28):285310. doi: 10.1088/0957-4484/22/28/285310 [17] KWOK H, BRIGGS K, TABARD-COSSA V. Nanopore fabrication by controlled dielectric breakdown [J]. PLoS One,2014,9(3):e92880. doi: 10.1371/journal.pone.0092880 [18] YANAGI I, ISHIDA T, FUJISAKI K, et al. Fabrication of 3-nm-thick Si3N4 membranes for solid-state nanopores using the poly-Si sacrificial layer process [J]. Scientific Reports,2015,5(1):1-13. doi: 10.9734/JSRR/2015/14076 [19] VENTA K, SHEMER G, PUSTER M, et al. Differentiation of short, single-stranded DNA homopolymers in solid-state nanopores [J]. Acs Nano,2013,7(5):4629-4636. doi: 10.1021/nn4014388 [20] SAWAFTA F, CARLSEN A T, HALL A R. Membrane thickness dependence of nanopore formation with a focused helium ion beam [J]. Sensors,2014,14(5):8150-8161. doi: 10.3390/s140508150 [21] LARKIN J, HENLEY R, BELL D C, et al. Slow DNA transport through nanopores in hafnium oxide membranes [J]. Acs Nano,2013,7(11):10121-10128. doi: 10.1021/nn404326f [22] GARAJ S, HUBBARD W, REINA A, et al. Graphene as a subnanometre trans-electrode membrane [J]. Nature,2010,467(7312):190-193. doi: 10.1038/nature09379 [23] MERCHANT C. DNA translocation through graphene nanopores [J]. Biophysical Journal,2010,100(3):521a. doi: 10.1016/j.bpj.2010.12.3046 [24] YUAN Z, LIU Y, DAI M. Controlling DNA translocation through solid-state nanopores [J]. Nanoscale Research Letters,2020,15(1):80. doi: 10.1186/s11671-020-03308-x [25] IVANOV A P, INSTULI E, MCGILVERY C M, et al. DNA tunneling detector embedded in a nanopore [J]. Nano Letters,2011,11(1):279-285. doi: 10.1021/nl103873a [26] WANG Y, SADAR J, TSAO C W, et al. Nanopore chip with self-aligned transverse tunneling junction for DNA detection [J]. Bisosensors and Bioelectronics,2021,193:113552. doi: 10.1016/j.bios.2021.113552 [27] GRAF M, LIU K, SARATHY A, et al. Transverse detection of DNA in a MoS2 nanopore [J]. Biophysical Journal,2018,114(3):180a. doi: 10.1016/j.bpj.2017.11.1005 [28] XIE P, XIONG Q, FANG Y, et al. Local electrical potential detection of DNA by nanowire-nanopore sensors [J]. Nature Nanotechnology,2012,7(2):119-125. doi: 10.1038/nnano.2011.217 [29] LI W, ZHOU J, MACCAFERRI N, et al. Enhanced optical spectroscopy for multiplexed DNA and protein-sequencing with plasmonic nanopores: Challenges and prospects [J]. Analytical Chemistry,2022,94(2):503-514. doi: 10.1021/acs.analchem.1c04459 [30] LANGECKER M, PEDONE D, SIMMEL F C, et al. Electrophoretic time-of-flight measurements of single DNA molecules with two stacked nanopores [J]. Nano Letters,2011,11(11):5002-5007. doi: 10.1021/nl2030079 [31] LING X S. DNA sequencing using nanopores and kinetic proofreading [J]. Quantitative Biology,2020,8(3):187-194. doi: 10.1007/s40484-020-0201-x [32] LIU X, SKANATA M M, STEIN D. Entropic cages for trapping DNA near a nanopore [J]. Nature Communications,2015,6:6222. doi: 10.1038/ncomms7222 [33] PEDONE D, LANGECKER M, MUENZER A M, et al. Fabrication and electrical characterization of a pore-cavity-pore device [J]. Journal of Physics-Condensed Matter,2010,22(45):454115. doi: 10.1088/0953-8984/22/45/454115 [34] 叶佳佳. 基于纳米孔测序的磁镊微控系统设计与制造 [D]. 南京: 东南大学, 2018.YE Jiajia. Design and fabrication of magnetic tweezers system based on nanopore sequencing [D]. Nanjing: Southeast University, 2018. [35] YANG H, WEI S, JI A, et al. Double layer nanopore fabricated by FIB and TEM [C]// IEEE. Proceedings of the 2017 IEEE International Conference on Manipulation, Manufacturing and Measurement on the Nanoscale (3M-NANO). Shanghai, 2017: 274-277. [36] LAM M H, BRIGGS K, KASTRITIS K, et al. Entropic trapping of DNA with a nanofiltered nanopore [J]. Acs Applied Nano Materials,2019,2(8):4773-4781. doi: 10.1021/acsanm.9b00606 [37] PEDONE D, LANGECKER M, ABSTREITER G, et al. A pore-cavity-pore device to trap and investigate single nanoparticles and DNA molecules in a femtoliter compartment: Confined diffusion and narrow escape [J]. Nano Letters,2011,11(4):1561-1567. doi: 10.1021/nl104359c [38] HARMS Z D, HAYWOOD D G, KNELLER A R, et al. Single-particle electrophoresis in nanochannels [J]. Analytical Chemistry,2015,87(1):699-705. doi: 10.1021/ac503527d [39] CHOI J, JIA Z, RIAHIPOUR R, et al. Label-free identification of single mononucleotides by nanoscale electrophoresis [J]. Small,2021,17(42):2102567. doi: 10.1002/smll.202102567 [40] ATHAPATTU U S, RATHNAYAKA C, VAIDYANATHAN S, et al. Tailoring thermoplastic in-plane nanopore size by thermal fusion bonding for the analysis of single molecules [J]. Acs Sensors,2021,6(8):3133-3143. doi: 10.1021/acssensors.1c01359 -





 下载:
下载:








 下载:
下载:








 邮件订阅
邮件订阅 RSS
RSS
